|
Resumen:
|
[ES] España es el segundo país en cuanto a área agrícola cultivada en la Unión Europea y se posiciona como el primer país con superficie de viñedo a nivel mundial, con más de novecientas mil hectáreas cultivadas en toda ...[+]
[ES] España es el segundo país en cuanto a área agrícola cultivada en la Unión Europea y se posiciona como el primer país con superficie de viñedo a nivel mundial, con más de novecientas mil hectáreas cultivadas en toda la península. Por lo tanto, el sector vitivinícola español tiene gran relevancia a nivel mundial.
La normativa de la Unión Europea obliga a la regulación fitosanitaria de los productos vegetales que son introducidos en los países miembros. La legislación española, así mismo, prohíbe la introducción y propagación en el país de virus y organismos afines, infecciosos para variedades de Vitis, y obliga al control de aquellos fitopatógenos sobre los que ya se tiene constancia. Por lo que es necesario controlar los patógenos que afectan a estos cultivos a fin de evitar pérdidas de producción y de calidad del producto vegetal final. En este sentido, los virus de mayor relevancia económica para el cultivo de la vid son Grapevine fleck virus (GFkV), Grapevine virus A (GVA), Grapevine virus B (GVB), Grapevine leafroll-associated virus 1 (GLRaV-1), Grapevine leafroll-associated virus 2 (GLRaV-2), Grapevine leafroll-associated virus 3 (GLRaV-3), Grapevine fanleaf virus (GFLV), Strawberry latent ringspot virus (SLRSV), Raspberry ringspot virus (RPRSV), Arabis mosaic virus (ArMV) y Tomato black ring virus (TBRV).
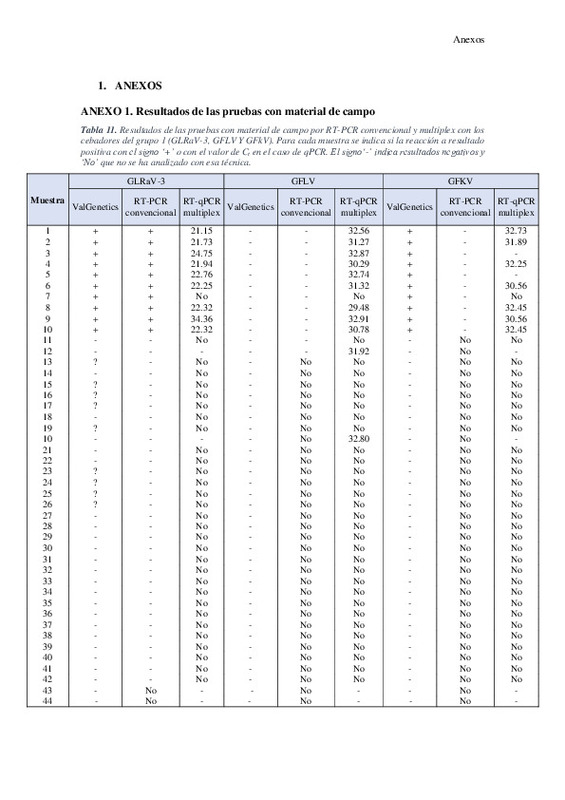
En este proyecto, se pretende desarrollar una metodología de PCR para la detección múltiple de los principales virus que afectan a vid, anteriormente mencionados, en una misma muestra vegetal. Para ello se diseñaron cebadores específicos de regiones conservadas entre cepas de la misma especie viral, como son los genes que codifican para las proteínas de la cápsida, así como sondas específicas de cada especie viral marcadas con diferentes fluoróforos. De este modo, se diseñaron “in silico” parejas de cebadores que teóricamente pudieran amplificar en base a sus características moleculares (porcentaje de citosina y guanosina, formación de homodímeros y heterodímeros…) y, además, se establecieron las condiciones de reacción de PCR óptimas. Una vez elegidos los cebadores y las condiciones de la PCR-multiplex ideales, se procedió a realizar las pruebas experimentales con los controles (positivo, negativo, de la extracción del ARN) para optimizar todo el proceso. En una última fase, se aplicó la metodología desarrollada al análisis fitopatológico de muestras vegetales de campo, procedentes de diferentes lugares y variedades de vid, para comprobar que la metodología funcionaba adecuadamente.
El objeto del presente proyecto es, por tanto, el desarrollo de una tecnología de PCR-multiplex para el análisis de los principales virus patógenos de la vid. Con el fin de diagnosticar su presencia una misma muestra, reduciendo el número, coste y tiempo de los análisis y así mejorar las técnicas de diagnóstico, detección y saneamiento empleadas en ValGenetics SL.
[-]
[EN] Spain is the second country with highest agricultural cultivated area in the European Union and it is positioned as the first country in large cultured vineyard surface worldwide, with more than nine hundred thousand ...[+]
[EN] Spain is the second country with highest agricultural cultivated area in the European Union and it is positioned as the first country in large cultured vineyard surface worldwide, with more than nine hundred thousand hectares cultivated. Therefore, the Spanish wine sector is economically important at international level.
The phytosanitary directive of the European Union controls the sanitary state of plant products that are introduced in or move between the member countries. The Spanish law also prohibits the introduction and spread of phytopathological viruses and other infectious organisms in Vitis, thus it forces to the control of those viral species. In this way, it is necessary to detect these pathogenic viruses in order to avoid losses in grape production and in the final product quality. On this regard, the viruses of major economic relevancy infecting grapevine varieties are Grapevine fleck virus (GFkV), Grapevine virus A (GVA), Grapevine virus B (GVB), Grapevine leafroll-associated virus 1 (GLRaV-1), Grapevine leafroll-associated virus 2 (GLRaV-2), Grapevine leafroll-associated virus 3 (GLRaV-3), Grapevine fanleaf virus (GFLV), Strawberry latent ring spot virus (SLRSV), Raspberry ringspot virus (RPRSV), Arabis mosaic virus (ArMV) and Tomato black ring virus (TBRV).
To reduce the number, cost and time of analysis, there is a need in detecting several viruses in a single genetic analysis. In this project, three PCR-multiplex tests have been developed in order to detect the above indicated principal grapevine viral species. For this aim, specific primers of conserved genomic regions, like capsid protein coding genes, were designed, as well as specific TaqMan probes, labeled with different dyes, targeting each of the viral species studied. Each couple of primers, was designed on the basis of its molecular characteristics (percentage of cytosine and guanine, self-dimers and hetero-dimer formation…) and the optimum PCR conditions were tested ¨in silico¨. Once the primers sequences and the optimum PCR-multiplex conditions were established, the experimental PCR-multiplex tests were initially performed with controls (positive, negative and ARN extraction) to optimize the whole analytical process. Afterwards, the developed PCR-multiplex methodology was applied for the analysis of field samples from different places and distinct varieties of grapevine, to verify the methodology.
The objective of the present project is, therefore, the development of a PCR-multiplex's technology to analyse the most important viruses affecting grapevine in three single analyses. In this work sampling, number, cost and time of the phytopathological analyses could be reduced and the diagnostic technologies and detection in plant sanitation experiments applied in ValGenetics SL improved.
[-]
|